神戸大学大学院理学研究科の安居佑季子研究員 (現・京都大学助教)、石崎公庸准教授らと、京都大学大学院生命科学研究科・河内孝之教授、信州大学理学部・久保浩義教授、近畿大学生物理工学部・大和勝幸教授、マックスプランク植物育種学研究所・Klaus Theres博士らの共同研究グループは、陸上植物の共通祖先に近いコケ植物ゼニゴケ※1を用いて、植物体から新たな芽をもつ独立したクローン個体を増殖させるための重要な因子を同定することに成功しました。さらに今回発見した因子は、被子植物の芽を増やす働きをもつ因子と共通する起源をもつことも明らかとなりました。
今回の発見は、植物が生涯、芽を増やし続ける仕組みの起源が、コケ植物と被子植物が別れた4億年以上前に遡ることを示唆しています。今後、この因子が関わる芽を増やす仕組みを更に解析することで、農業や園芸分野において、様々な植物を効率よく増産させる技術の改良に貢献できると期待されます。
この研究成果は、11月7日 (現地時間) に、米国の学術誌「Current Biology」に掲載されました。
ポイント
- コケ植物ゼニゴケで、体細胞クローン繁殖体である無性芽を新生する場を作るのに必須な遺伝子としてGCAM1を発見しました。
- GCAM1遺伝子がコードするタンパク質をゼニゴケで過剰に機能させると、幹細胞※2の性質を持つ未分化状態の細胞が増殖しました。
- GCAM1遺伝子は、植物に広く保存されているR2R3-MYB型転写因子をコードしており、被子植物の同じ種類の遺伝子は腋芽※3形成に機能することが知られています。
- コケ植物の無性芽と被子植物の腋芽は無性的に芽を増殖する点で共通しており、本研究の成果は、植物の増殖システムの基本原理とその進化の道筋の解明に貢献します。
研究の背景
植物は生涯を通して成長点である芽、幹細胞を持ち続け、葉や花などの器官分化を繰り返します。また多くの植物は、幹細胞の維持のみならず、新たに生み出すことができます。クローン個体を介した無性生殖もその例で、腋芽が地下部の塊茎となるジャガイモ、地上部の腋芽が独立して脱離するヤマイモのムカゴや、葉の周縁部から新たな植物体ができるコダカラベンケイソウがこれにあたり、これらの繁殖様式は栄養繁殖と呼ばれています。栄養繁殖で生み出されるクローン個体は、元個体と同じ遺伝型を持つため、効率よく増殖させることで、優れた形質をもつ個体を安定して増産することが可能になります。このため、植物の栄養繁殖は農業や園芸分野で広く利用されていまが、その仕組みには不明な点が多く残されています。
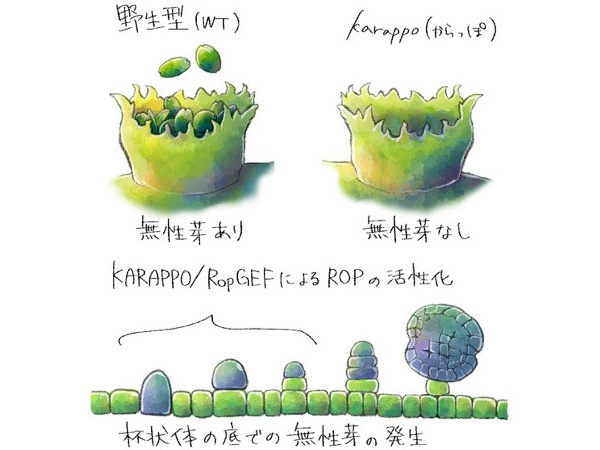
本研究グループは、植物進化の基部に位置するコケ植物ゼニゴケに着目して、研究を進めています。ゼニゴケは、非常に繁殖力が強い植物ですが、その理由の一つとして、クローン繁殖体である無性芽を大量に作ることが挙げられます (図1)。ゼニゴケの無性芽は、体の表面にできる杯状体という器官の中で作られます。最近、本研究グループによって、杯状体の中で無性芽の形成が開始される分子メカニズムが明らかにされました。しかし、無性芽ができる場が、どのようにしてできるかの分子メカニズムは全く分かっていませんでした。

A: ゼニゴケの杯状体。中に無性芽ができているのが見える。
B: 無性芽の拡大写真。幹細胞としての性質を持つメリステムを両側に持つ。矢印はメリステムを指す。
C: 無性芽が葉状体に成長した写真。成長点であるメリステムが分岐して成長していく。
研究の内容
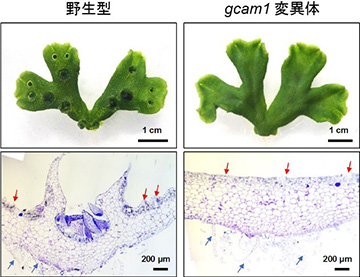
上段: 生育3週間の植物体
下段: 杯状体断面の切片観察像。赤矢印は気室を、青矢印は腹鱗片を指す
本研究グループでは、網羅的な遺伝子発現解析により、杯状体で高く発現している遺伝子群を同定し解析していました。その中に含まれていた転写因子の1つに着目し、GEMMA CUP-ASSOCIATED MYB1 (GCAM1) と名付けました。GCAM1遺伝子を欠損させた変異体を作出したところ、この変異体では杯状体が全くできないことがわかりました (図2)。変異体において、杯状体以外の植物の成長は正常であったことから、GCAM1遺伝子がコードするGCAM1タンパク質は杯状体発生に特異的に機能する杯状体形成のマスター制御因子であることがわかりました。
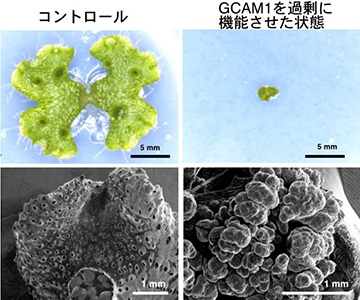
上段: 生育2週間の植物体
下段: 走査型電子顕微鏡での観察像
次に、GCAM1タンパク質が実際にどのような機能を持つことで、杯状体発生を制御しているかを調べるため、ゼニゴケの生体内でGCAM1を過剰に機能させることができる株を作出しました。GCAM1の機能が過剰になると、正常な植物の成長が起こらず、幹細胞としての性質を持つ未分化状態の細胞が増殖しました (図3)。このことから、野生型におけるGCAM1は、杯状体ができる領域において、幹細胞的な性質を維持し、杯状体と無性芽形成を制御していると考えられました。
GCAM1遺伝子はR2R3-MYB型転写因子をコードしていますが、被子植物の同じ種類の遺伝子 (シロイヌナズナ※4のRAXsやトマトのBlind) は腋芽形成に機能することが知られています。そこで、進化的な関係を調べるため、モデル被子植物であるシロイヌナズナを用いた解析を進めました。シロイヌナズナにおいてRAXs遺伝子を欠損させた変異体では、野生型に比べ腋芽の数が少なることが知られています。そこで、この変異体にゼニゴケのGCAM1遺伝子を導入したところ、腋芽の数が回復しました。このことは、ゼニゴケのGCAM1遺伝子が、シロイヌナズナにおいて、腋芽形成に機能し得ることを示しており、GCAM1とシロイヌナズナの遺伝子がオーソログ※5の関係にあることがわかりました (図4)。
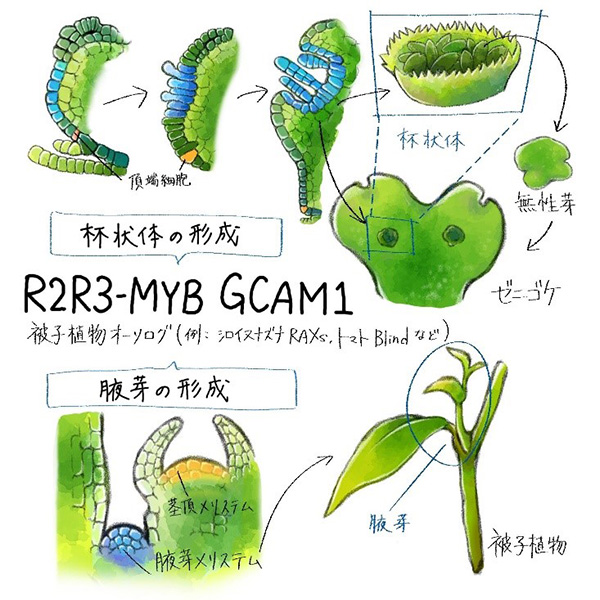
GCAM1はゼニゴケの杯状体形成に、被子植物のオーソログである因子は、腋芽形成に機能する。杯状体内にできる無性芽と腋芽はどちらも無性的に芽を増殖する点で共通している。
今後の展開
ゼニゴケのGCAM1遺伝子と被子植物の腋芽形成を制御する遺伝子がオーソログの関係にあったことから、ゼニゴケの杯状体と被子植物の腋芽には共通の仕組みがある可能性が考えられます。
多くの主要作物は被子植物であり、その腋芽形成の制御技術は、農作物の増産に直接的に関わります。今後、GCAM1が関わる遺伝子制御のネットワークを調べ、被子植物との共通性を明らかにする事で、ゼニゴケの栄養繁殖にとどまらず、植物が新たな芽を増産する共通の基本メカニズムが解明され、農産物を効率よく生産する技術の基盤となることが期待されます。
用語解説
- ※1 ゼニゴケ
- コケ植物タイ類に属する (学名Marchantia polymorpha)。2017年に全ゲノム配列が解読され、実験室での培養が容易、遺伝子導入や遺伝子改変が容易などの理由から、新たなモデル植物として注目されている。
- ※2 幹細胞
- 自己複製能力と様々な細胞に分化する能力を持つ特殊な細胞のこと。
- ※3 腋芽
- 茎と葉の間に形成されるの芽のこと。植物によっては枝分かれした茎の基部から発根するため新たなクローン個体の形成と捉えることができる。
- ※4 シロイヌナズナ
- 被子植物アブラナ科の一年草 (学名Arabidopsis thaliana)。2000年に全ゲノム配列が解読され、モデル植物として植物研究の材料に広く使われている。
- ※5 オーソログ
- 進化上、種を超えて共通の祖先型遺伝子を持つと考えられる遺伝子であり、種分化により派生した相同遺伝子のこと。
謝辞
本研究は神戸大学を中心に、京都大学、信州大学、マックスプランク植物育種学研究所、近畿大学の協力により行われました。また、本研究は以下の研究助成を受けて行われました。
- 科学研究費新学術領域研究 (No. 18H04836, 25113009, 25119711, 15H01233, 17H06472)
- 科学研究費基盤研究 (B)(No. 15H04391, 19H03247)
- 科学研究費若手研究 (No. 19K16167)
- 基礎生物学研究所 共同利用研究
- 旭硝子財団研究助成
- サントリー生命科学財団 SUMBOR GRANT
論文情報
- タイトル
- “GEMMA CUP-ASSOCIATED MYB1, an Ortholog of Axillary Meristem Regulators, Is Essential in Vegetative Reproduction in Marchantia polymorpha”
- DOI
- 10.1016/j.cub.2019.10.004
- 著者
- Yukiko Yasui,1,2# Shigeyuki Tsukamoto,1,# Tomomi Sugaya,3 Ryuichi Nishihama,2 Quan Wang,4 Hirotaka Kato,1 Katsuyuki T. Yamato,5 Hidehiro Fukaki,1 Tetsuro Mimura,1 Hiroyoshi Kubo,3 Klaus Theres,4 Takayuki Kohchi,2 and Kimitsune Ishizaki1*
1 神戸大学大学院理学研究科
2 京都大学大学院生命科学研究科
3 信州大学理学部
4 マックスプランク植物育種学研究所
5 近畿大学生物理工学部
#共同筆頭著者
*Corresponding author - 掲載誌
- Current Biology