神戸大学先端バイオ工学研究センターの秀瀬涼太特命教授、蓮沼誠久教授らの研究グループは、バイオものづくり(生物を活用して物質や製品を生産する技術)の生産性を高める有用酵素発見のための新技術を開発しました。①タンパク質のアミノ酸配列から酵素の性質を見分け、分類できる「MUSASHI(ムサシ)メソッド」の開発、②ラボラトリーオートメーション(ロボットを使って自動化する設備)を取り入れた酵素機能評価システムの構築-を行い、その組み合わせによる実験ワークフローを実現したものです。この新技術を活用し、未知活性を有する酵素や、既知の活性を凌駕する高活性酵素の識別にも成功しました。有用酵素の発見はバイオものづくりの推進に向けた最重要課題の一つであり、本研究の新技術は、バイオ資源の活用を基盤とする循環型経済社会(バイオエコノミー化)実現への貢献が期待できます。
この研究成果は、6月27日(金)に国際学術誌「ACS Catalysis」に掲載されました。
ポイント
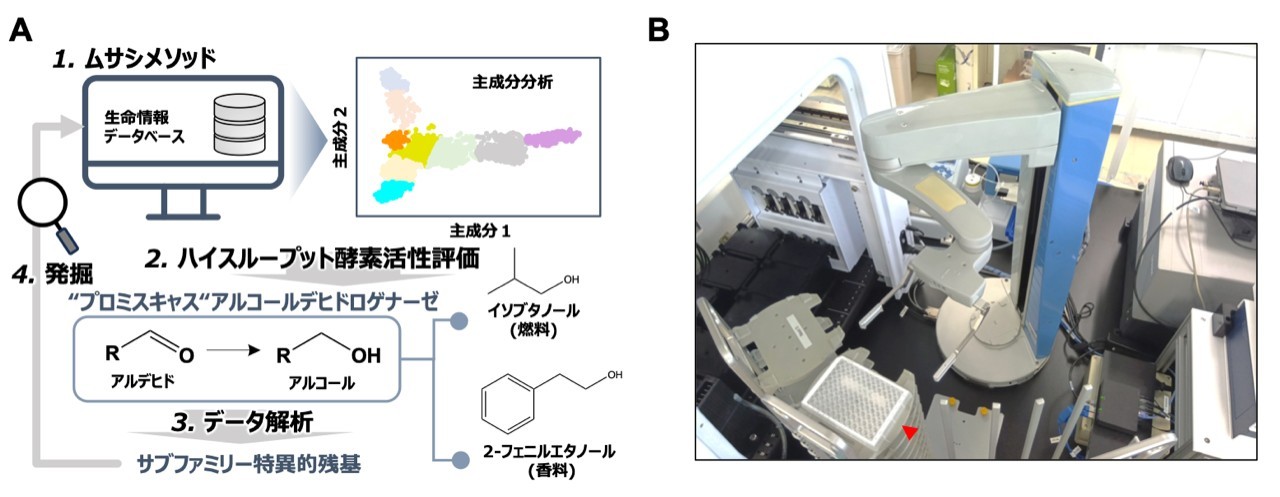
- タンパク質のアミノ酸配列(アミノ酸の並ぶ順番)をもとに、酵素の性質を見分けられる「MUSASHI(ムサシ)メソッド」(図1A)という新しい分類方法を開発しました。
- タンパク質の性能を高速かつ正確に評価するため、ラボラトリーオートメーションを実装した独自のハイスループット(酵素活性などを高速、自動的に評価する技術)システム(図1B)を構築しました。
- 上記の新手法、新システムを活用し、香料や燃料、プラスチックなどの原料として使用されるアルコールの生産に必要な酵素「アルコールデヒドロゲナーゼ」について、未知活性と高活性を決定する「配列的特徴」を見つけることに成功しました。
- 「ムサシメソッド」を活用することにより、タンパク質群の体系的データ取得が可能となり、有用酵素の探索を迅速かつ高効率にします。
研究の背景
地球温暖化や石油資源の枯渇が懸念される中、微生物の力で燃料や有用な化学物質をつくる技術が注目されています。こうした「バイオものづくり」は、従来の化学的手法とは異なり、常温・常圧といった穏やかな条件で反応を進められるため、大量のエネルギーを必要とせず、危険な有機物質なども使用せずに済みます。さらに、植物由来の糖や二酸化炭素といった再生可能な資源を活用するため、環境への負荷を抑えることができます。この分野の発展により、地球規模の課題の解決と経済成長を両立する社会の実現が可能になります。近年では、DNAの一部をピンポイントで書き換えるゲノム編集技術や、高速かつ高精度な化合物の分析技術など、バイオテクノロジーの急速な進歩により、食品や医薬品だけでなく、石油から製造されていた化学素材や燃料などの原料を、より短時間、低コストで生産できるようになってきました。
こうした「バイオものづくり」においては、使用する酵素が非常に重要です。酵素は、生物の体内でさまざまな化学反応を助ける存在であり、物質の生産を実現する決め手となります。従来は、酵素を選ぶ際、すでに実験で性質が明らかになっているものを使うのが一般的でした。しかし、未知の酵素を活用することができれば、より一層効率的に多種多様な有用物質を生み出せる可能性があります。そのための情報源となるのが、世界中から集められた生命情報が格納されている公共のデータベース(米国NIHが運営しているNCBIなど)です。そこには、様々な生物が持つ酵素の情報が2億件以上も登録されており、現在も増加の一途を辿っています。ただし、その性質に関する情報の多くは推測されたものであり、実際の働きを確かめるには時間と労力がかかる実験を必要とします。
酵素の中には、本来の目的とは異なる物質にも働く「プロミスキャス酵素」が存在します。こうした酵素を活用すれば、1つの酵素で複数の有用物質を生産できる可能性が高まり、物質ごとに個別に酵素を選ぶ手間が省けて、開発のスピードアップにつながります。しかし、酵素がプロミスキャスかどうかは、酵素の「設計図(アミノ酸配列)」を見ただけでは簡単には分かりません。したがって、酵素の機能を正しく見抜くことは、バイオものづくりにおける大きな課題となっています。
研究の内容
神戸大学の研究チームは、香料や燃料、プラスチックの原料となる有用なアルコールの生産に重要な酵素であるADH(アルコールデヒドロゲナーゼ)※1を研究対象に定め、以下に示す新技術を開発しました。
1. ムサシメソッドによる酵素分類
公共データベースに登録されている6,727件のADH配列データをコンピューターに読み込ませ、配列間の類似性と違いを数値化して分析しました。その際、独自の真理値表※2を用いて、酵素の配列情報を2次元の地図のように可視化しました。この結果、性質が似ている酵素をグループ化することに成功し、この酵素分類法を「MUSASHI(MUltiple-Sequence Alignment-based protein Selection via clustering using HIgh-dimensional analysis:ムサシ)メソッド」と名付けました。
2. ラボラトリーオートメーションを利用したハイスループット酵素活性評価
酵素活性を効率よく測定するため、ラボラトリーオートメーション技術を活用した新たな評価システムを構築しました。このシステムは、試薬の分注装置、光の検出機器、小型実験プレート搬送ロボットから構成され、~0.1 ml程度の反応系で、アルコール生成時の光の変化を検出して、活性を計算します。このシステムにより、1日あたり2,000回以上の測定が可能となり、従来の人手による測定と比べて4倍以上の処理能力(スループット)を達成しました。
具体的には、ムサシメソッドで分類された各グループから代表的な酵素を選抜し、41種類の市販アルデヒドを用いて活性を調べました。その結果、従来よく使用されているADHよりも5倍以上高い生産効率で、イソブタノールや2-フェニルエタノールといった有用なアルコールの生産に成功しました。
3. 上記2つを組み合わせた実験ワークフローで酵素の特徴的なアミノ酸配列を同定
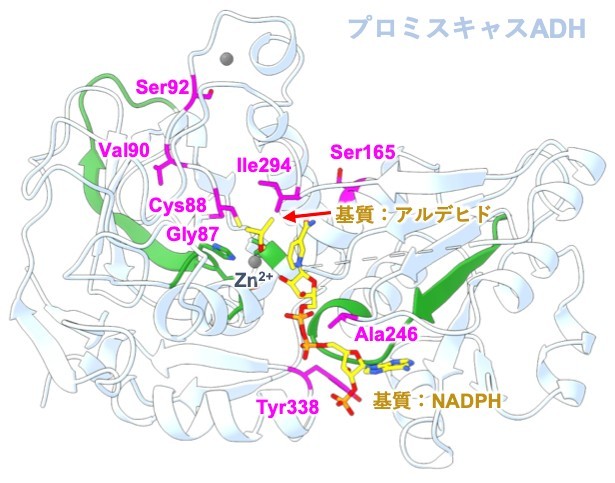
ムサシメソッドによる酵素分類結果と、ハイスループット酵素活性評価で得られた酵素活性データをもとに、プロミスキャスADHに共通する特徴的な配列(サブファミリー特異的残基)を同定しました(図2)。このサブファミリー特異的残基は、今後ますます拡大するデータベースの中から、有望な酵素をすばやく見つけ出すための新たな指標として活用されることが期待されます。
今後の展開
「酵素を制する者が、バイオものづくりを制する」と言って過言ではありません。本研究の「有用酵素を発掘する新しい技術」は、バイオものづくりの基盤技術として活用されることが期待されます。バイオものづくりにおいて、プロミスキャス酵素を効果的に活用するためには、目的とする物質に対して高い選択性と生産効率を持たせるような酵素の改変(エンジニアリング)が不可欠です。プロミスキャス酵素は、人工的に有望な酵素を作り出す出発点として極めて有用であり、従来にはない新しい反応系の構築など、新規酵素の創出につながる可能性を大いに秘めています。
今後の研究においては、人工的に改変された有用酵素の開発や酵素機能を予測するAIモデルの構築といった重要課題にも、基礎的かつ重要なデータを提供する技術として貢献することが期待されます。
用語解説
※1 アルコールデヒドロゲナーゼ:
アルデヒドからアルコールへ変換する酵素。お酒を作る際に、酵母と呼ばれる微生物はこの酵素を使って、アルコール(エタノール)を生産する。
※2 真理値表:
0と1の並び(=ビット列)を使って、アルファベット文字で表示されるアミノ酸をコンピュータにわかる形で表現する表。
謝辞
本研究は、NEDO「植物等の生物を用いた高機能品生産技術の開発(スマートセルプロジェクト)」、NEDO「カーボンリサイクル実現を加速するバイオ由来製品生産技術の開発事業(バイオものづくりプロジェクト)」、JSPS「地域中核・特色ある研究大学強化促進事業(J-PEAKS)」の支援を受けて実施されました。
論文情報
タイトル
DOI
10.1021/acscatal.5c02764
著者
Ryota Hidese, Kanae Sakai, Musashi Takenaka, Keiji Fushimi, Hisashi Kudo, Kenya Tanaka, Ryo Nasuno, Christopher J. Vavricka, Akihiko Kondo, and Tomohisa Hasunuma.
掲載誌
ACS Catalysis