新潟大学農学部の山崎将紀教授、神戸大学のRym Fekih学術研究員、ユーロフィンジェノミクス株式会社の尾鼻孝浩マーケティングマネージャーらの共同研究グループは、これまで研究が難しいと考えられていた日本のイネにおいてDNAの詳細な解析に成功しました。トヨタ自動車が開発したGRAS-Di(Genotyping by Random Amplicon Sequencing-Direct)と呼ばれる新技術は再現性や正確性が高く、低コストかつ簡便に実施でき、日本のイネの遺伝解析における技術革新になるとともに、他の植物だけでなく、動物や微生物への応用が期待できます。また、現在新潟大学が養成している、大規模な日本のイネ実験集団を使った新品種開発の推進も期待されます。
本研究成果は、2023年2月17日、科学誌「Plants」にEarly Access Versionとして掲載されました。
ポイント
- GRAS-Di技術は日本のイネの遺伝子型決定に有効で、再現性や正確性が高く、低コストかつ簡便に実施でき、他の生物への応用が期待できる。
- GRAS-Di技術により、日本のイネを使った解析が加速し、詳細な遺伝解析が可能になった。
研究の背景
日本のイネは比較的近縁なためにDNA塩基配列が似ており、品種や系統間のDNAの違い(DNA多型)と遺伝子型(注1)を検出することがこれまで容易ではなく、コストも高いことで研究の進捗が制限されていました。最近のDNA検出技術の発展はめざましく、ヒトだけでなく植物へも適用されています。本研究グループは、GRAS-Di(Genotyping by Random Amplicon Sequencing-Direct)技術について、日本のイネ品種や系統の遺伝子型を低コストに検出できるように技術検証を進めて、ゲノム全体を網羅する連鎖地図(注2)の作成を目指してきました。
日本のイネは北海道から九州・沖縄まで栽培されており、開花期などの重要な形質に多様性が観察されます。この日本のイネの多様性の全容、つまり関与する遺伝子の同定と各品種における遺伝的効果を解明することも目的に研究を進めてきました。
研究の概要と成果
(1)
日本のイネ品種や系統の遺伝子型を低コストに検出できるように技術開発を進めました。使用したイネは食用米品種「コシヒカリ」と酒米品種「山田錦」を交雑して世代を進めた集団 (190系統で構成) と両親である「コシヒカリ」と「山田錦」です。全DNA配列を決定できる次世代シークエンサーと呼ばれる機器を使いました。
GRAS-Di技術は以下のとおりです。63種のプライマーを高濃度と低温処理により、ゲノムDNA全体をPCRによって増幅された断片を次世代シークエンサーでDNA配列を決定して、DNA多型と遺伝子型を検出できる簡便で再現性の高い手法です (図1参照)。
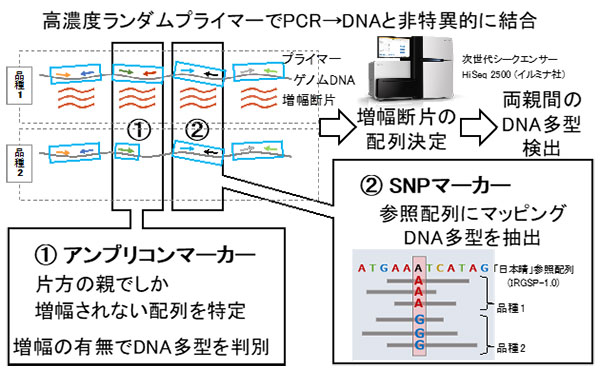
GRAS-Di(Genotyping by Random Amplicon Sequencing-Direct)技術をイネに適用した。
(GRAS-Di:1,050箇所、SNP遺伝子型:272箇所)
全DNA配列を決定するためにはゲノムの30倍程度のDNA配列を解読する必要があります。「コシヒカリ」と「山田錦」との交雑集団を使った結果、GRAS-Di技術はイネゲノムの1倍でも十分な遺伝子型が得られ、1,050箇所の遺伝子型を決定できました。これで低コスト化が実現できました。この集団はすでにSNP遺伝子型 (272箇所) がわかっており、GRAS-Di技術が3.8倍を超える遺伝子型を決定できました。GRAS-Di技術による遺伝子型とSNP遺伝子型を比較して、正確性を調べたところ99.7%を超えており、GRAS-Di技術の信頼度が確認できました。
(2)
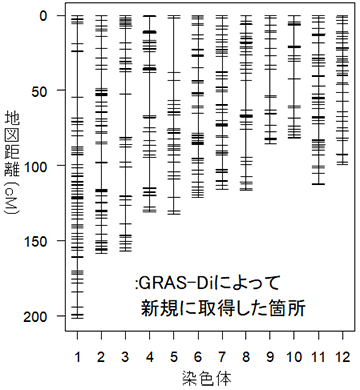
「コシヒカリ」と「山田錦」との交雑集団を使って、ゲノム中の相対位置を示す連鎖地図を作成しました。GRAS-Di技術 (1,050箇所) とSNP遺伝子型 (272箇所) を統合した結果高密度な連鎖地図ができ、GRAS-Di技術により新規に配置した箇所が増えました (図2参照)。これまで地図距離の平均5.3cMに対して、平均2.9cMへと改善しました。
(2021年兵庫県加西市のデータを使った。)
(3)
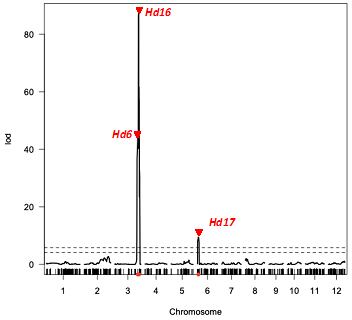
「コシヒカリ」と「山田錦」交雑集団と連鎖地図を使って、開花期の遺伝解析を行いました。開花期は2021年に兵庫県加西市で栽培して調査したものです。出穂期遺伝子であるHd6, Hd16, Hd17が検出されました (図3参照)。GRAS-Di技術により、詳細な遺伝解析ができました。
(4)
その他にも集団について解析し、GRAS-Di技術が高精度な遺伝子データを数多く検出しました。
- 「コシヒカリ」と「台中65号」交雑集団 (96系統) GRAS-Di:1,258箇所、SNP遺伝子型:272箇所 (約4.6倍増)。連鎖地図の地図距離平均5.5cM→3.4cMに改善。
- 「コシヒカリ」と「藤坂5号」交雑集団 (95系統) GRAS-Di:1,708箇所、SNP遺伝子型:262箇所 (約6.5倍増)。連鎖地図の地図距離平均5.8cM→3.0cMに改善。
- 「コシヒカリ」と「双葉」交雑集団 (94系統) GRAS-Di:1,704箇所、SNP遺伝子型:266箇所 (約6.4倍増)。連鎖地図の地図距離平均4.8cM→3.4cMに改善。
今後の展開
GRAS-Di技術は簡便で再現性が高く、本研究によりイネ1サンプル数千円で解析できることがわかり、従来の手法だと1サンプルあたり数万円から比べると大幅なコストダウンができるようになりました。この技術は日本のイネの遺伝解析に革新をもたらしており、他の植物だけでなく、動物や微生物への応用が期待できます。
現在「コシヒカリ」と29品種を交雑して世代をすすめた集団 (総計3,268系統) が養成されており、GRAS-Di技術による遺伝子型決定や連鎖地図作成を進めています。また、開花期だけでなく、収量性も調査して遺伝解析を進めています。この集団の中から新たな新品種開発が期待されており、現在その開発資金獲得のためのクラウドファンディングを公開し、ご支援を募っています (3/31まで)。
用語解説
- (注1) DNA多型と遺伝子型、SNP
- DNA多型とはDNA塩基配列の違いで生じる変異です。例えば、DNAを構成するアデニン (A)、シトシン (C)、グアニン (G)、チミン (T) による違い (SNP: Single Nucleotide Polymorphism、一塩基多型) やDNAの挿入や欠失もあります。ここで遺伝子型とはDNA多型を比較・識別して、遺伝子領域の構成を示します。ある子孫が両親 (ここではイネ品種親Xとイネ品種親Yを想定) から受け継いだ遺伝子構成を明らかにし、親Xホモ型、ヘテロ型、親Yホモ型を識別します。
- (注2) 連鎖地図
- 遺伝子型のデータから、ゲノム中の相対的位置を直線上に示したものです。これを基礎として遺伝解析が行われることが多いです。地図距離をcM (センチモルガン) と表します。
謝辞
本研究は、科学技術振興機構戦略的創造研究推進事業「環境変動に対する植物の頑健性の解明と応用に向けた基盤技術の創出」(JPMJCR17O3)、総合科学技術・イノベーション会議のSIP(戦略的イノベーション創造プログラム)「次世代農林水産創造技術」(生研センター)の支援を受けて行われました。
論文情報
- タイトル
- “High-Density Linkage Maps from Japanese Rice japonica Recombinant Inbred Lines Using Genotyping by Random Amplicon Sequencing-Direct (GRAS-Di)”
- DOI
- 10.3390/plants12040929
- 著者
- Rym Fekih1, Yohei Ishimaru1, Satoshi Okada1,2, Michihiro Maeda1, Ryutaro Miyagi3, Takahiro Obana3, Kazuyo Suzuki4, Minoru Inamori4, Hiroyuki Enoki4 and Masanori Yamasaki1,5
1: 神戸大学大学院農学研究科附属食資源教育研究センター、2: 名古屋大学、3: ユーロフィンジェノミクス株式会社、4: トヨタ自動車株式会社、5: 新潟大学大学院自然科学研究科 - 掲載誌
- Plants