神戸大学先端バイオ工学研究センターの加藤俊介准教授、蓮沼誠久教授、工藤恒特命助教らと、大阪大学大学院工学研究科の林高史教授らの研究グループは、主成分分析を活用した大規模データ解析手法により、生物が本来行わない「非天然反応」を、分子の立体構造を厳密に制御しながら実行する新規酵素の発見に成功しました。今後、持続可能な化学合成や創薬、機能性材料の開発に資する新しいバイオ触媒探索基盤としての応用が期待されます。
この研究成果は、2026年1月28日にAngewandte Chemie International Edition 誌にオンライン掲載されました。

ポイント
- 主成分分析※1を基盤とする大規模データ解析手法により、データベース上に存在するタンパク質の配列情報を体系的に解析することで、生物が本来行わない「非天然反応」を高立体選択的に触媒できる新規酵素※2を発見しました。
- これまで自然機能予測に用いられてきたデータ解析手法を、「非天然反応」という新しい枠組みへと展開することで、従来のタンパク質工学とは異なるアプローチによるバイオ触媒開発の新たな方向性を示しました。
- 本研究により、同一の化学反応に対して異なる立体構造の生成物を選択的に作り分ける酵素群を同定でき、今後の持続可能な化学合成や創薬、機能性材料の開発への応用が期待されます。
研究の背景
酵素は、生体内で起こる化学反応を高い効率と選択性で進めることができるため、医薬品や化学品の製造、環境に優しいものづくりなど、さまざまな分野で注目されています。さらに近年では、生物が本来行わない人工的な化学反応「非天然反応」を酵素に担わせる研究も進められており、従来の化学触媒に代わる持続可能な化学プロセスとして期待されています。
これまで、このような非天然反応を触媒する酵素を得るためには、特定の酵素を出発点として、アミノ酸配列を人工的に改変する「タンパク質工学」や「指向性進化※3」といった手法が主に用いられてきました。しかし、この方法では探索が特定の酵素配列の周辺に限定されるため、自然界がタンパク質に与えた広大な配列空間※4のごく一部しか調べることができないという課題がありました。
一方で、近年のゲノム解析技術の急速な進展により、データベース上には膨大な数のタンパク質配列情報が蓄積されています。これらの配列データを体系的に解析するためのデータ解析手法(主成分分析など)は、これまで主に「その酵素が生物の中でどのような自然機能を持つか」を予測する目的で使われてきました。しかし、これらの手法が、生物が本来行わない「非天然反応」を担う酵素の探索に有効であるかどうかは検証されておらず、その可能性自体が不明でした。
そこで本研究では、従来は自然機能予測に用いられてきたデータ解析手法を非天然反応という新しい枠組みへと展開し、自然界に存在する多様な酵素配列の中から、有望なバイオ触媒を効率的に見いだすことをめざしました。本研究には、人工的な配列改変に頼らず、自然界の配列多様性そのものを活用して新しい触媒機能を発掘できるのではないか、という期待がありました。
研究の内容
本研究では、自然界に存在する膨大な数の酵素配列情報に着目し、それらを体系的に解析することで、生物が本来行わない「非天然反応」を触媒できる酵素を見いだすことをめざしました。特に、スチレンとジアゾ酢酸エチルという2つの化学物質からシクロプロパン化合物を合成する反応を触媒できる微生物酵素の探索を行いました。この反応は、通常は金属触媒を用いた化学合成で行われるものであり、生物が自然に行う反応ではありません。さらにこの反応では、生成物として4種類の異なる立体構造を持つシクロプロパン化合物(立体異性体)が生じるため、それらを選択的に作り分けることが重要な課題となります。これまでに、Frances Arnold博士らの研究グループによって、人工的に改変されたヘム依存性酵素※5、6がこの反応を触媒できることは報告されていましたが、4種類の異なる立体異性体を選択的に作り分ける酵素を体系的に見いだした例はありませんでした。
そこで本研究では、まず初めに、微生物のゲノムデータベースに登録されている膨大な数のヘム依存性酵素、特にグロビン※7と呼ばれるタンパク質ファミリーを対象に、主成分分析に基づくデータ解析手法「Musashiメソッド」を用いた網羅的な探索を行いました。この解析により、データベース上のグロビンが、配列情報の特徴に基づいて2次元平面に可視化・分類され、それらが非天然反応に対し高い触媒活性や立体選択性を示す可能性があることを予想しました(図1)。研究グループは、この解析結果をもとに有望な酵素候補を選び出し、実際の反応評価を行いました。その結果、自然界に存在する複数の微生物のグロビンが、スチレン(化合物1)とジアゾ酢酸エチル(化合物2)の立体選択的なシクロプロパン化反応を触媒できることが明らかとなり、さらに、Musashiメソッドで分類された酵素群ごとに異なる立体構造の生成物を選択的に作り分けることが分かりました(図2)。これは、自然界の酵素配列多様性を活用することで、分子の立体構造を厳密に制御しながら、4種類すべての立体異性体(化合物(R,R)-3, (S,S)-3, (S,R)-4, (R,S)-4)を選択的に与える立体多様(stereodivergent)合成が可能であることを示した成果です。
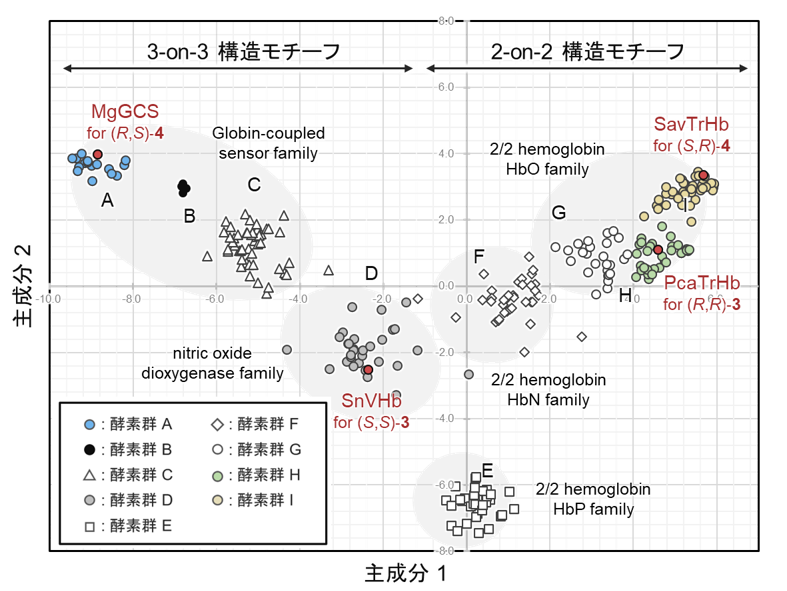
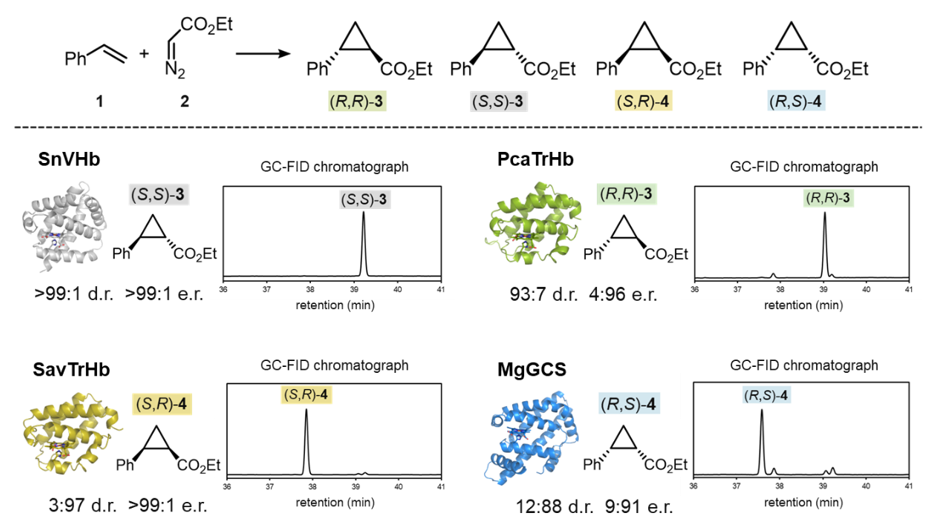
図2:本研究で同定したシクロプロパン化合物の立体多様(stereodivergent)合成を実現する酵素
さらに本研究では、酵素探索に加えて、なぜ特定の酵素が異なる立体異性体を選択的に生成できるのかを理解するために、配列データの統計解析を用いました。特に、立体選択性の異なる酵素群同士のアミノ酸を比較することで、生成物の立体構造を決定するうえで重要と考えられるアミノ酸残基や立体構造モチーフを同定しました。さらに、これらの解析結果に基づいて実際にアミノ酸配列を改変した酵素を作製したところ、生成物であるシクロプロパン化合物の立体選択性が設計通りに変化することを確認し、統計解析による予測を実験的に立証しました。
本研究では、従来は生物の自然機能予測に用いられてきたデータ解析手法を、非天然反応という新しい応用対象へと展開し、自然界に存在する酵素配列多様性そのものを活用した新しいバイオ触媒探索の可能性を示すことに成功しました。人工的なタンパク質改変に依存せず、大規模データ解析や統計解析を起点として高い触媒活性と立体選択性を兼ね備えた酵素を見いだせる本手法は、今後、持続可能な化学合成や創薬、機能性材料開発などへの幅広い応用が期待されます。
今後の展開
本研究で示された手法は、自然界に存在する膨大な数の酵素配列情報を大規模データ解析によって体系的に整理し、これまで見過ごされてきた触媒機能や、自然界には無い触媒機能を効率的に発掘できる新しい研究基盤となります。特定の酵素を出発点として人工的に改変する従来の方法とは異なり、自然界の配列多様性そのものを活用し、より広大なタンパク質配列空間を探索できる点が、本手法の大きな特長です。このアプローチを応用することで、今回対象としたシクロプロパン化反応に限らず、これまで酵素では実現が難しいと考えられてきたさまざまな非天然反応に対しても、有望なバイオ触媒を見いだせる可能性が広がります。今後、人工知能(AI)やデータ解析技術を組み合わせた酵素探索が一層加速し、将来的には、医薬品や農薬、機能性材料などの合成において、環境負荷の低い持続可能な化学プロセスの実現に貢献することが期待されます。
用語解説
※1 主成分分析
多数の情報を含むデータから重要な特徴を抽出し、全体の関係性を分かりやすく整理・可視化する解析手法です。本研究では、タンパク質配列の特徴をまとめることで、膨大な数の酵素を俯瞰的に分類しました。
※2 酵素
化学反応を触媒する機能を持つ生体分子(タンパク質)の総称です。
※3 指向性進化
生物の進化の仕組みを実験室内で模倣し、タンパク質の性能を向上させるタンパク質工学手法の1つです。具体的には、タンパク質にランダムな変異を導入し、その中から目的に適した変異体を選び出す操作を繰り返しながら、タンパク質の性能を向上させます。
※4 配列空間
タンパク質を構成する20種類のアミノ酸の並び(配列)の組み合わせを、空間として捉えた概念です。自然界には、アミノ酸配列が少しずつ異なる膨大な数のタンパク質が存在しており、配列空間はそれらの多様性の広がりを表しています。
※5 ヘム
鉄原子を含む生体分子で、タンパク質の中に組み込まれることで、酸素運搬や化学反応に重 要な役割を果たします。
※6 ヘム依存性酵素
酸素の運搬や化学反応を進めるためにヘムを必要とする酵素の総称です。
※7 グロビン
ヘムと結合する特徴的な立体構造(グロビンフォールド)を持つタンパク質群の総称で、酸素の結合や感知などに関与します。代表例として哺乳類のヘモグロビンやミオグロビンが有名ですが、微生物にも多様なグロビンが存在します。
謝辞
本研究は、科学技術振興機構(JST) 戦略的創造研究推進事業 ACT-X(JPMJAX22B6)、日本学術振興会(JSPS) 科学研究費助成事業(学術変革領域研究(A)公募研究:JP25H01579(予知生合成)、JP24H01136(超越分子システム)、JP23H04554(予知生合成)、JP22H05421(超越分子システム)、基盤研究(B):JP24K01630、若手研究:JP22K14783、研究活動スタート支援:JP21K20535、基盤研究(A):JP25H00887、国際共同研究加速基金(国際先導研究):JP22K21348)の支援を受けて実施されました。
また、本研究の一部は、一般財団法人 蛋白質研究奨励会 金子・成田研究奨励金ならびに第一三共株式会社「はばたく次世代」応援寄付プログラムの支援を受けて行われました。ここに記して深く感謝いたします。
論文情報
タイトル
DOI
10.1002/anie.202526025
著者
Shunsuke Kato*, Koki Takeuchi, Kohei Umeda, Hisashi Kudo, Tomohisa Hasunuma, Takashi Hayashi*
掲載誌
Angewandte Chemie International Edition
報道問い合わせ先
神戸大学総務部広報課
E-Mail:ppr-kouhoushitsu[at]office.kobe-u.ac.jp(※ [at] を @ に変更してください)